Asocian el parásito Anisakis con cáncer de colon
7 agosto, 2024
El Grupo de Investigación en Hematología del HCU Lozano Blesa realiza la primera aféresis para terapia CART en Aragón
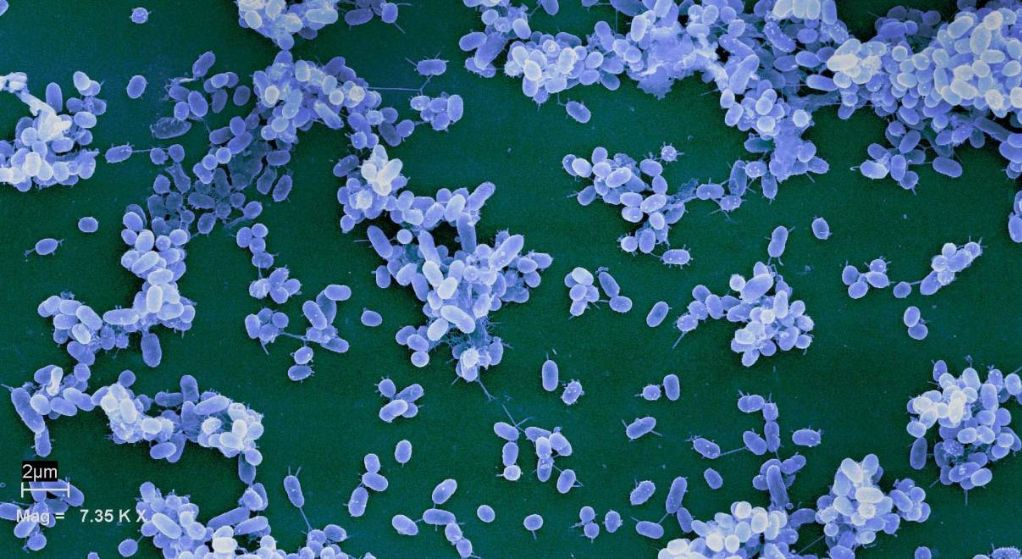
12 agosto, 2024Un estudio liderado por científicos de la Estación Experimental del Zaidín (CSIC) ha identificado un superfamilia de proteínas que incluye más de 6.300 receptores muy extendidos entre las bacterias. El avance podría abrir nuevas estrategias para combatir a las patógenas
La búsqueda de nuevas herramientas para combatir bacterias patógenas es una prioridad a nivel mundial. Las bacterias necesitan adaptarse a diferentes entornos para poder sobrevivir y, por ello, emplean receptores bacterianos con el fin de ajustar su fisiología y garantizar su supervivencia en entornos cambiantes.
Un equipo de la Estación Experimental del Zaidín (EEZ-CSIC), en colaboración con el Laboratorio de Estudios Cristalográficos del Instituto Andaluz de Ciencias de la Tierra (IACT-CSIC) y la Ohio State University (EE UU), ha identificado una gran familia de receptores bacterianos con características comunes.
Concretamente, han comprobado que tienen la capacidad de unirse a las purinas, un tipo de moléculas orgánicas, mediante un patrón específico conservado en las proteínas bacterianas. Estas purinas incluyen productos de la degradación de ácidos nucleicos y compuestos vegetales como la cafeína y la teofilina.
Este significativo avance, publicado en la revista Nature Communications, podría abrir nuevas vías para el desarrollo de métodos para combatir las infecciones bacterianas, al interferir en estos receptores.
«Una estrategia para combatir bacterias patógenas es bloquear las señales que activan su virulencia, evitando que infecten y dañen al huésped»
Una de las estrategias que se utilizan para combatir bacterias patógenas consiste en bloquear las señales que disparan la virulencia, es decir, evitar que las bacterias activen los mecanismos que les permiten infectar y dañar al huésped.
Este proceso se basa en intervenir en el funcionamiento de los receptores de estas bacterias. Sin embargo, la falta de conocimiento sobre las señales que activan los receptores bacterianos dificulta el desarrollo de métodos efectivos para bloquearlos, y, por tanto, impedir la virulencia.
La importancia de los ligandos
Mediante una combinación de técnicas de biología estructural, bioinformática, bioquímica de proteínas y microbiología, los autores de este estudio muestran que la superfamilia identificada incluye más de 6.300 receptores que se encuentran muy extendidos en numerosas especies bacterianas, incluyendo aquellas que son patógenos de humanos y plantas.
Asimismo, estos cuentan con roles cruciales en la regulación de la actividad genética, el metabolismo celular, la señalización interna y la capacidad de movimiento de las bacterias.
Los investigadores utilizaron la bacteria Vibrio cholerae, agente causante del cólera, como modelo en su estudio, demostrando que la unión de estos receptores a compuestos como la teofilina, una purina abundante en el té, aumenta los niveles de un segundo mensajero que controla la virulencia.
«Nuestro enfoque novedoso permitirá predecir señales en otras familias de dominios y tendrá un gran impacto en microbiología»
Tino Krell (EEZ-CSIC)
Este trabajo abre así una nueva vía de estudio para combatir patógenos, interfiriendo con su capacidad de detectar señales externas. Del mismo modo, se consigue validar un innovador método para identificar y clasificar grandes grupos de proteínas bacterianas que se unen a compuestos denominados ligandos.
Además, se demuestra que los derivados de las purinas son importantes señales químicas externas que influyen en las funciones bacterianas, abriendo nuevas posibilidades de tratamiento de este tipo de infecciones.
“El enfoque que hemos utilizado en el estudio es novedoso y permitirá en el futuro predecir las señales identificadas por otras familias de dominios, lo que tendrá un impacto importante en el campo de la microbiología”, indica Tino Krell, investigador de la EEZ-CSIC.
Referencia: Elizabet Monteagudo-Cascales, et al. «Ubiquitous purine sensor modulates diverse signal transduction pathways in bacteria». Nature Communications, 2024
Fuente: Agencia SINC
Imagen: EEZ-CSIC
Fuente: CSIC
Derechos: Creative Commons